AI位点预测服务是基于人工智能与结构生物信息学相结合的方法,识别蛋白质或其他生物大分子中的潜在功能位点,如小分子结合位点、催化位点、突变敏感区、抗原表位等。Neorabio融合深度学习模型与经典结构特征分析算法,提供高效、可靠的位点预测解决方案,助力早期药物靶点开发、分子设计及抗体筛选等研究方向。

服务流程
[提交结构] → [结构预处理] → [参数设置] → [对接运行] → [结果分析] → [报告交付]
应用场景
药物筛选前期靶点口袋识别与活性位点确认
结合位点筛选与结构修饰建议,支持分子对接与虚拟筛选
突变体设计及功能敏感区域分析
抗原表位预测用于抗体设计、疫苗开发等免疫研究
协助构建药效团模型或定向优化小分子结构
蛋白工程与合成生物学中的位点重构与功能增强
技术优势
基于深度学习与大规模蛋白结构训练的预测模型,准确率高
支持AlphaFold、PDB结构直接输入,快速适配新型蛋白靶标
综合考虑形状、极性、电荷、保守性等多维特征
结合分子模拟/对接等后续服务,提供一体化设计方案
可视化输出预测位点位置、评分与可信度等级
拥有经验丰富的生物信息与计算化学团队,保障预测质量与科学性
项目主要结果展示
蛋白质口袋(protein pocket)在生物分子学中扮演着关键角色,特别是在药物设计和分子相互作用研究中。蛋白质口袋是指蛋白质表面或内部的凹陷区域,这些区域通常是配体分子(如小分子药物、辅因子、底物等)结合的位点。无论在酶催化、信号传导、蛋白质-蛋白质相互作用等方面都具有重要作用。预测蛋白质口袋是药物设计中的重要步骤。通过确定潜在的药物结合位点,可以设计和优化小分子药物,以提高其特异性和有效性。精准的口袋预测有助于发现新的药物靶点,并加速药物开发过程。一般口袋预测的方法包含三种:基于几何形状的方法、基于能量的方法、以及基于机器学习的方法。这些方法结合使用可以显著提高预测的准确性,从而推进药物开发和分子功能研究。
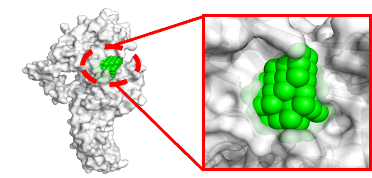
PLI位点预测结果
交付内容
蛋白活性位点;口袋氨基酸组成
交付内容










